R1a – YDNA Wenedów, Sklawenów i Lachów [Archiwum 03 2018]
R1a – YDNA Wenedów, Sklawenów i Lachów [Archiwum 03 2018]
Opracowanie powstało na podstawie bazy danych – wyników badań YDNA w projekcie FTDNA R1a-Project. Jest on w zasadzie dla innego celu – szukanie krewnych, przodków przy pomocy badań DNA. Tym niemniej ma ponad 7500 członków z haplogrupą R1a (marzec 2018). Organizacja tych danych jest niestaranna i wymaga pracy z ich „wyczyszczeniem”, aby były przydatne do opracowania statystyki. Na końcu przydatne okazało się niecałe 4 tysiące wyników. Aby otrzymać wyniki w pełni satysfakcjonujące należałoby wykonać badania statystyczne mężczyzn w Europie w ilości około 15-20 tysięcy próbek i to z jak największą dokładnością subcladów. Tym niemniej duży błąd statystyczny przy 4 tysiącach próbek dotyczy niektórych krajów o mniejszej liczbie mieszkańców oraz niższych haplogrup (młodszych mutacji), gdzie może sięgać +/- 30%.
Ponieważ dane w projekcie FTDNA nie są próbkami reprezentatywnymi dla poszczególnych państw, zastosowano odpowiednie przeliczniki ilościowe i proporcjonalne, podobnie jak dla analizy haplogrupy dynarskiej. Przyjąłem założenie, że dane Eupedii dotyczące częstotliwości występowania R1a w poszczególnych państwach są poprawne.
Celem pracy jest dostarczenie danych o charakterze statystycznym, które mogą być przydatne dla osób badających historie Słowian. Zarówno amatorsko jak i zawodowo. Otrzymane wyniki konfrontowałem z różnymi źródłami i uznałem za zadowalające. Co ciekawe w wielu przypadkach podważają dane w tzw. źródłach oficjalnych, szczególnie w sytuacji, gdy te źródła są starsze niż 10 lat oraz powstały na zdecydowanie mniejszej liczbie próbek. W efekcie wiele dotychczasowych informacji, w tym mapy z rozmieszeniem haplogrup dostarczają niepełne dane lub nawet niepoprawne, a wyciągane na ich podstawie wnioski są wątpliwe.
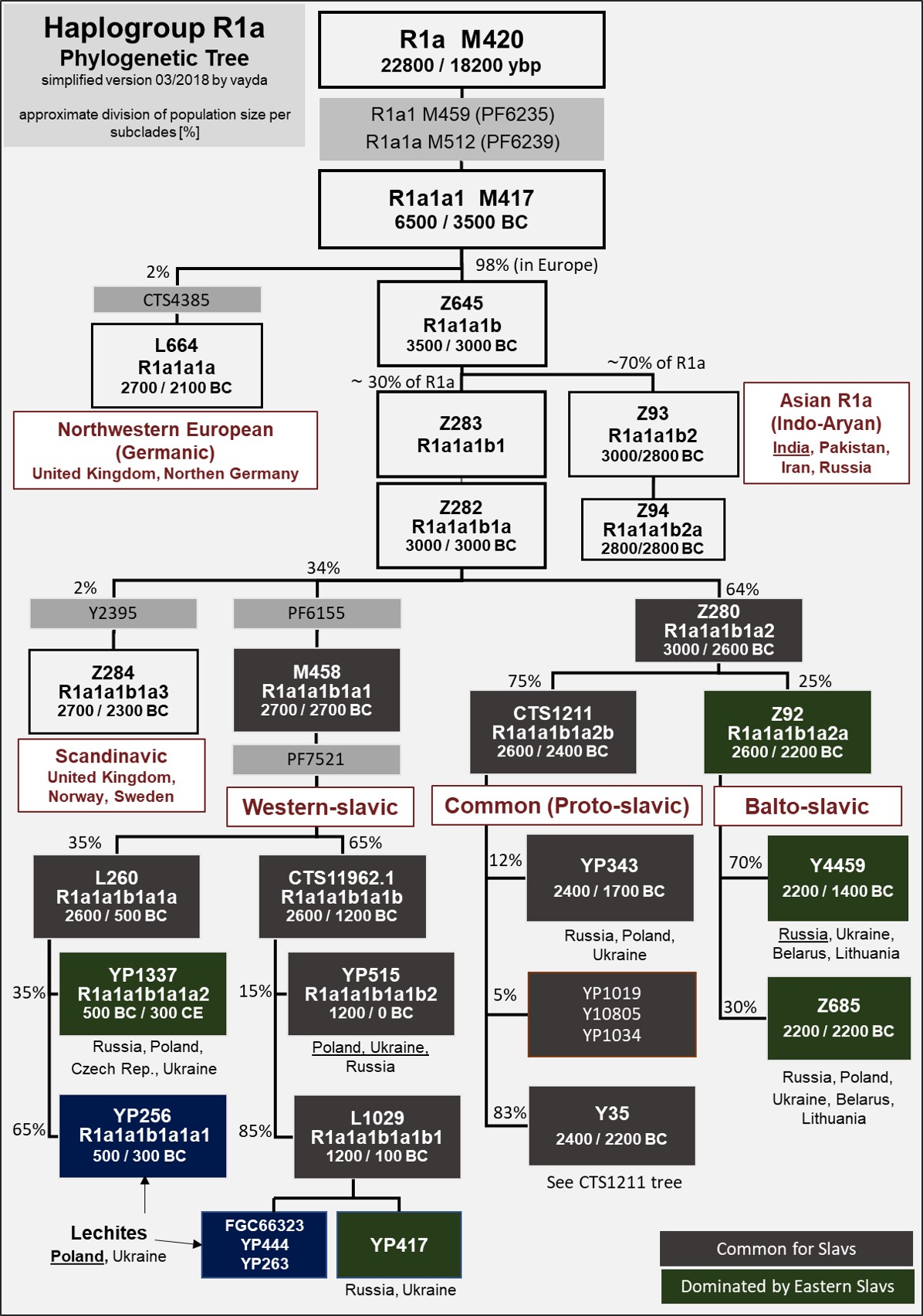
Poniższe drzewo mutacji R1a zawiera zebrane informacje o haplogrupach, czasie ich powstania (formed/TMRCA), proporcjach populacji dla niższych subcladów oraz rejony występowania.
Haplogrupa R1a określana jest jako Indo-Europejska. W Europie dominujące są subclady Z280 oraz M458, stanowiące łącznie prawie 90% populacji R1a na tym kontynencie. Ich wspólny przodek o mutacji Z282 żył około 3000 lat p.n.e (formed/ TMRCA 5000 lat temu). Jest również przodkiem mutacji skandynawskiej Z282. Obecnie mężczyzna u którego wystąpiła mutacja Z282 ma 70 milionów męskich potomków.
Poza Europą R1a występuje w Azji – głównie mutacja Z93. Przyjmując udział w populacji takich krajów jak Indie i Pakistan 25-30%, Iran 15%, Iran 5%, można szacować Z93 na około 200 mln mężczyzn w Azji. Ta haplogrupa praktycznie nie występuje w Europie, a można ją szacować na kilkaset tysięcy mężczyzn. Tym niemniej uważa się, powstała wg różnych poglądów w Europie na terenie Polski (Kujawy) i Rosji lub Turcji, a potem emigrowała do Indii i Persji.
Tabela poniżej zawiera główne haplogrupy R1a oraz wielkość ich populacji.
| Ethnos/Region | ISOGG clasification | Markers | Formed/TMRCA [ybp] | Population [mln] | Distribution |
| Common Slavic | R1a1a1b1a2b | Z280>CTS1211 | 4600 / 4400 | 34,2 | 44% |
| Western Slavs | R1a1a1b1a1 | M458 | 4700 / 4700 | 23,4 | 30% |
| Eastern Slavs (Balto-Slavic) | R1a1a1b1a2a | Z280>Z92 | 4600 / 4200 | 11,3 | 14% |
| Scadinavian | R1a1a1b1a3 | Z284 | 4700 / 4300 | 3,0 | 4% |
| Northwestern European | R1a1a1a | L664 | 4700 / 4100 | 1,6 | 2% |
| Asian (Indo-Aryan) | R1a1a1b2 | Z93 | 5000 / 4800 | 0,3 | 0,4% |
| others | 4,2 | 5% | |||
| TOTAL | 78,0 | 100% | |||
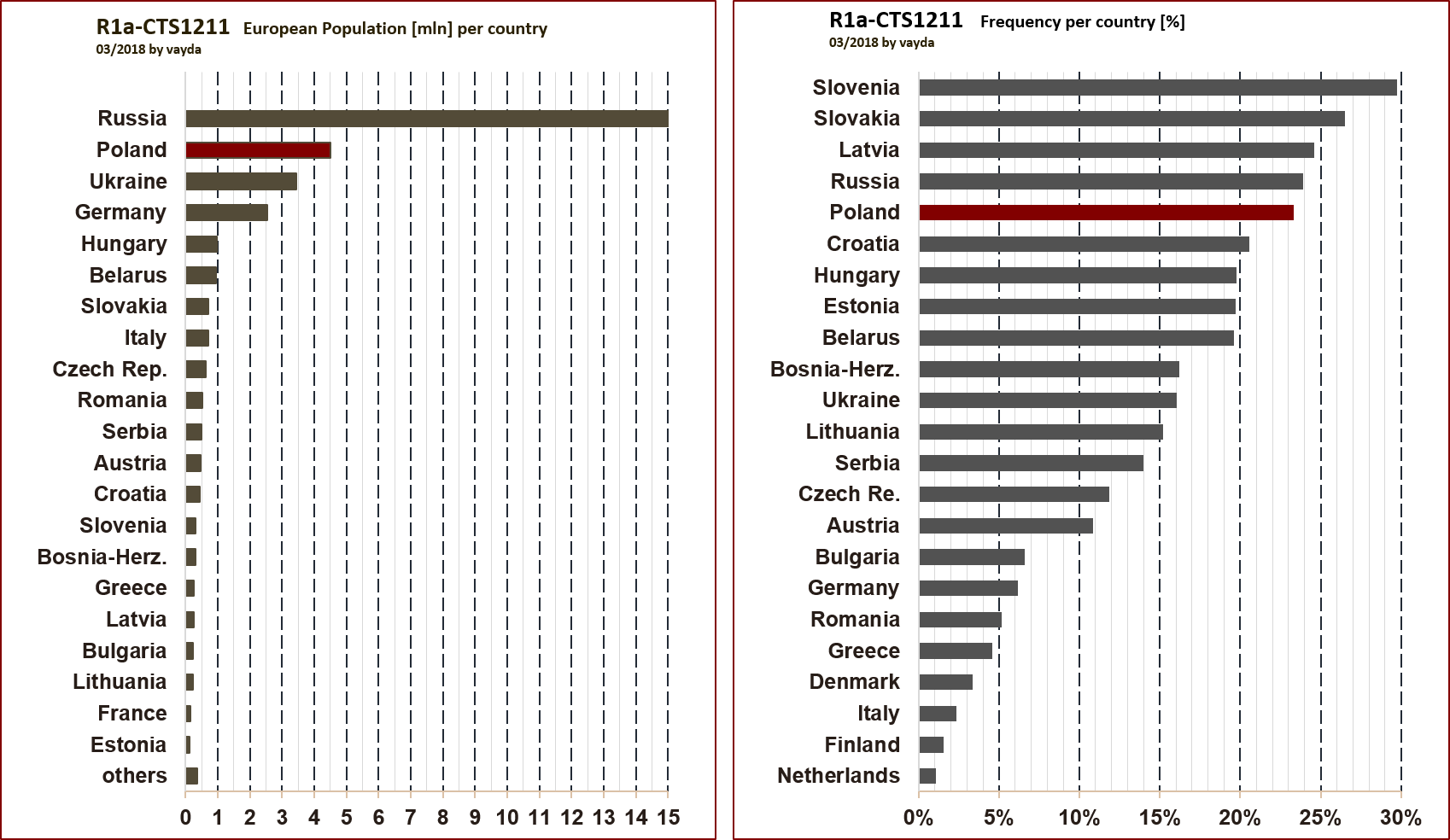
Tabela poniżej pokazuje częstotliwość występowania haplogrup R1a w Europie.
R1a – frequency of major sublades per country [% of male population] 03/2018 by vayda
| Country | R1a [mln] | R1a [%] | CTS1211 | M458 | Z92 | Z284 | L664 | others | Samples |
|---|---|---|---|---|---|---|---|---|---|
| Europe | 78 mln | 21.6% | 9.5% | 6.5% | 3.1% | 0.8% | 0.4% | 0.013 | 4317 |
| Poland | 11.1 | 57.5 | 23.3 | 27.8 | 4.9 | 0 | 0.1 | 1.4 | 764 |
| Belarus | 2.4 | 51 | 19.6 | 17 | 13.1 | 0 | 0 | 1.3 | 93 |
| Russia | 29.2 | 46 | 23.9 | 8.9 | 10.3 | 0.3 | 0 | 2.6 | 721 |
| Ukraine | 9.4 | 44 | 16.1 | 19.8 | 6.3 | 0.3 | 0 | 1.5 | 204 |
| Slovakia | 1.1 | 41.5 | 26.5 | 11.5 | 3.5 | 0 | 0 | 0 | 50 |
| Latvia | 0.4 | 40 | 24.6 | 9.2 | 3.1 | 0 | 3.1 | 0 | 18 |
| Lithuania | 0.6 | 38 | 15.2 | 11.4 | 10.1 | 0.6 | 0 | 0.7 | 81 |
| Slovenia | 0.4 | 38 | 29.7 | 5 | 1.7 | 0 | 1.7 | 0 | 27 |
| Czech R. | 1.7 | 33 | 11.9 | 18 | 1 | 0.5 | 1 | 0.6 | 66 |
| Estonia | 0.2 | 32 | 19.7 | 4.9 | 4.9 | 2.5 | 0 | 0 | 13 |
| Moldova | 0.5 | 30.5 | 0 | <10 | |||||
| Hungary | 1.5 | 29.5 | 19.8 | 7.9 | 0.9 | 0 | 0.3 | 0.6 | 109 |
| Norway | 0.7 | 25.5 | 0.3 | 0.3 | 0 | 21.5 | 1.9 | 1.5 | 322 |
| Croatia | 0.5 | 24 | 20.6 | 3.4 | 0 | 0 | 0 | 0 | 16 |
| Iceland | 0 | 23 | 0 | <10 | |||||
| Austria | 0.8 | 19 | 10.9 | 7.5 | 0 | 0 | 0 | 0.6 | 34 |
| Bosnia-Herz. | 0.3 | 18 | 16.2 | 1.2 | 0.6 | 0 | 0 | 0 | 32 |
| Romania | 1.8 | 18 | 5.1 | 10.3 | 2.6 | 0 | 0 | 0 | 19 |
| Serbia | 0.6 | 18 | 12.4 | 4.4 | 1.3 | 0 | 0 | 0 | 385 |
| Bulgaria | 0.6 | 17 | 6.6 | 8.2 | 1.6 | 0 | 0 | 0.6 | 34 |
| Germany | 6.6 | 16 | 6.2 | 6.4 | 0.7 | 0.3 | 1 | 1.4 | 345 |
| Sweden | 0.8 | 16 | 1.1 | 1.2 | 0.2 | 10.1 | 1.6 | 1.8 | 203 |
| Denmark | 0.4 | 15 | 3.3 | 2.5 | 0.8 | 6.7 | 1.7 | 0 | 24 |
| Macedonia | 0.1 | 13.5 | 0 | <10 | |||||
| Greece | 0.6 | 11.5 | 6 | 3 | 1,6 | 0 | 0 | 1 | 12 |
| Albania | 0.1 | 9 | 0 | <10 | |||||
| Scotland | 0.2 | 8.5 | 0.4 | 0.1 | 0.1 | 7.2 | 0.8 | 0 | 148 |
| Montenegro | 0.02 | 7.5 | 0 | <10 | |||||
| Finland | 0.1 | 5 | 1.6 | 1.4 | 0.6 | 1 | 0.3 | 0.1 | 96 |
| England | 1.2 | 4.5 | 0.1 | 0.2 | 0.1 | 2.1 | 1.5 | 0.5 | 249 |
| Belgium | 0.2 | 4 | 0 | <10 | |||||
| Italy | 1.2 | 4 | 2.3 | 0.4 | 0.3 | 0 | 0 | 1 | 50 |
| Netherlands | 0.3 | 4 | 1.1 | 0 | 0 | 0.4 | 1.8 | 0.7 | 17 |
| Switzerland | 0.1 | 3.5 | 0.6 | 1 | 0 | 0.3 | 0.6 | 1 | 11 |
| France | 1 | 3 | 0.4 | 0.9 | 0 | 0.4 | 0.4 | 0.9 | 21 |
| Ireland | 0.1 | 2.5 | 0.2 | 0 | 0 | 1.1 | 0.7 | 0.5 | 106 |
| Spain | 0.5 | 2 | 0 | <10 | |||||
| Portugal | 0.1 | 1.5 | 0 | <10 | |||||
| N. Ireland | 0.01 | 1 | 0 | 0 | 0 | 0.8 | 0.2 | 0 | 16 |
Sources: Eupedia (R1a frequency [%]); FTDNA R1a-Project (R1a samples).; https://dnk.poreklo.rs (additional R1a samples for Serbians – 358)
Z tabeli wynika, że najwyższą zgodność etnosu narodowego z haplogrupą R1a ma Polska i Białoruś – wynik powyżej 50%. W Europie wyższy udział konkretnej haplogrupy w populacji mają jedynie Walia (74%), Hiszpania (60%) i Francja (58,5%) dla R1b.
Haplogrupy „niesłowiańskie”:
Haplogrupa Z284 określana jest jako skandynawska i najczęściej na mapach umieszcza się ją w Norwegii ze względu na najwyższy jej udział (25% mężczyzn w tym kraju). Tym niemniej porównywalna liczebnie jest w Anglii i Szwecji.
Haplogrupa L664 określana jest jako północno-zachodnia lub germańska i tradycyjnie lokowana w północnych Niemczech. Niesłusznie, bowiem taka sama liczba Z664 jest w Anglii.
Haplogrupa Z93 Indo-Aryjska jest praktycznie nieobecna w Europie.
Haplogrupy R1a słowiańskie to Z280 (CTS1211 oraz Z92) i M458.
Utożsamianie tych haplogrup ze Zachodnimi czy Wschodnimi Słowianami jest dużym uproszczeniem, a w niektórych przypadkach błędne. Haplogrupa M458 jest powszechnie określana jako zachodnio-słowiańska, a Z280 (CTS1211+Z92) jako wschodnio-słowiańska. Inna sprawa to, że do Zachodnich Słowian zalicza się oficjalnie Polskę, Czechy, Słowację oraz zgermanizowanych Słowian. A dlaczego nie zachodnich Ukrainców i Białorusinów? Używając ten tradycyjny podział i nazewnictwo trudno jest opisacć obszary występowania haplogrup, zatem będę się posługiwać pojęciem zachodnich i wschodnich (pisane z małej litery) Słowian, gdzie granica przebiega na Dnieprze. Chodzi o podział geograficzny, ale mający też uzasadnienie historyczne chociażby jako granica Polski Jagiellonów. Również genetycznie. Żeby to wyjaśnić muszę podsumować pracę dotyczącą rozmieszczenia haplogrup Z280 i M458 i ich subcladów.
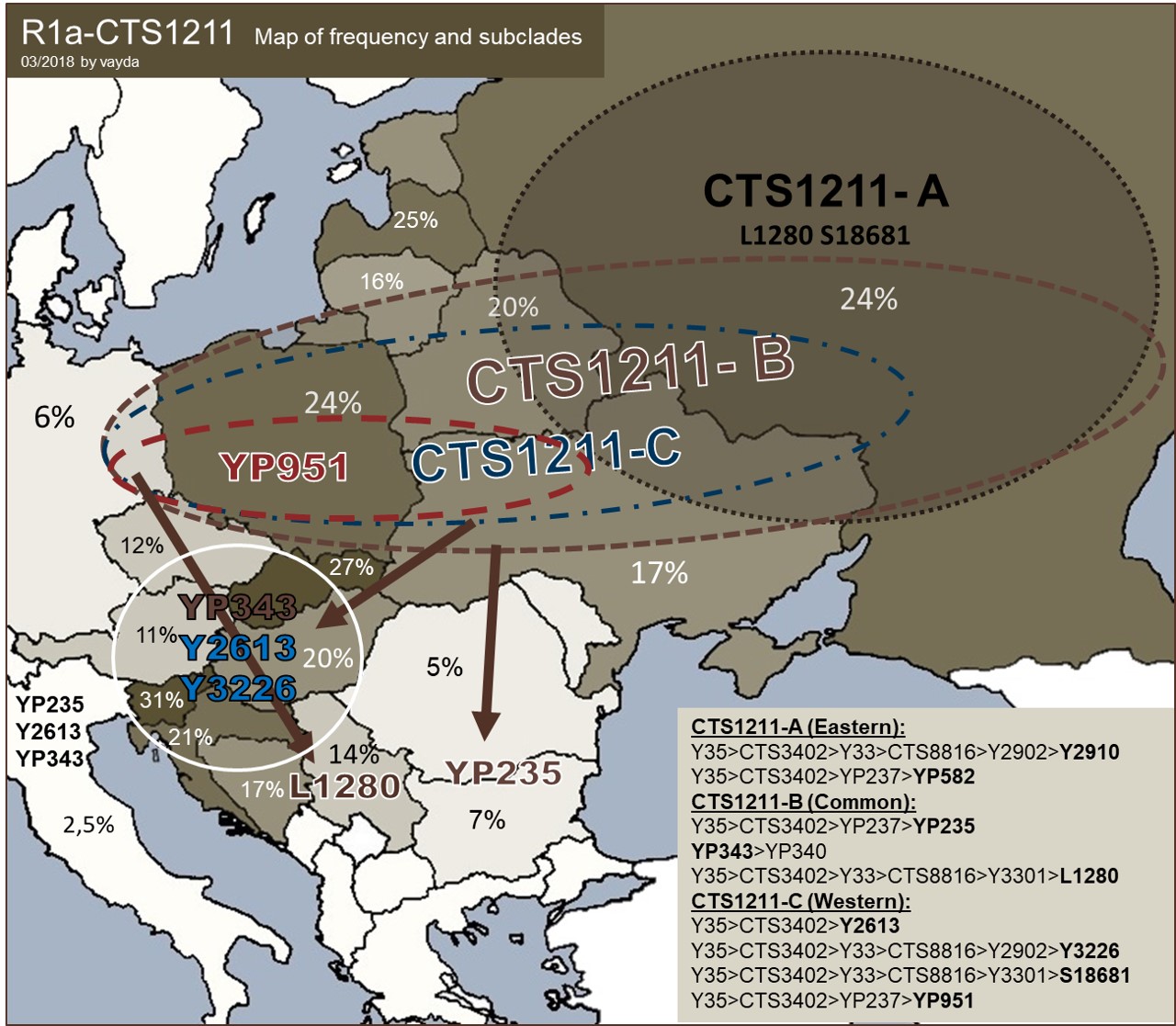
R1a-CTS1211
Haplogrupa CTS1211 jest największą populacją R1a (34 mln) i występuje powszechnie wśród Słowian. Przypisuje się ją do Wschodnich Słowian, ale to jest nieprawidłowe. Na tym poziomie mutacji sprzed 4 tysięcy lat (formed 4600, TMRCA 4400 lat temu) i ogromnej ruchliwości ludności Wschodniej Europy przez tysiące lat, przypisanie tej haplogrupy do zawężonego obszaru czy szczególnego etnosu jest błędem. Wśród obecnych narodów występuje z częstością 10-30%, przy czym w Polsce i Rosji wynosi około 24%.
więcej u źródła: http://blog.vayda.pl/r1a/
Czytajcie też u nas i na blogu Wayda.pl:
blog.wayda.pl: R1a – YDNA Wenedów, Sklawenów i Lachów (2018)