WYNIKI BADAŃ DNA ADRIANA LESZCZYŃSKIEGO
na podstawie projektu GENO 2.0 National Geographic 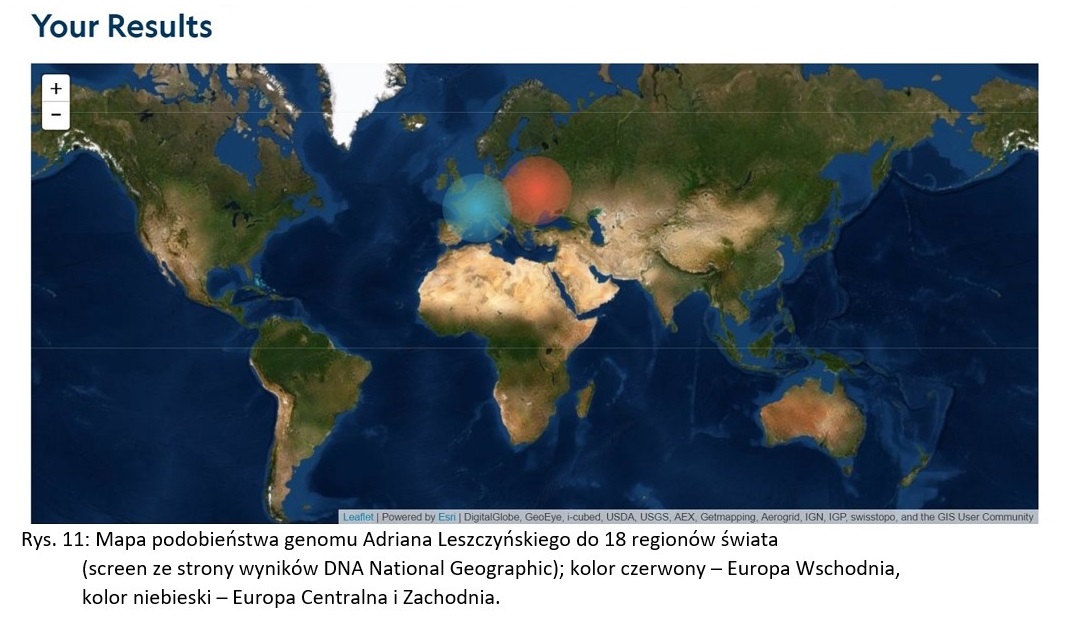
Pochodzenie ludności oraz historia od dawna mnie fascynują. Od pewnego czasu szczególnie pasjonuje mnie pochodzenie Słowian, a wśród nich Polaków. W obecnych czasach nie sposób mówić o pochodzeniu ludności, narodów i grup etnicznych bez uwzględnienia badań genetycznych. Sam również wiele razy zabierałem głos w sprawie tych badań. Napisałem na ten temat także kilka artykułów. W końcu zeszłego 2018 roku doszedłem do wniosku, że sam również powinienem zrobić sobie test DNA i zbadać swoje osobiste pochodzenie sięgając daleko wstecz.
Interesowało mnie jakie mam pochodzenie wedle haplogrupy męskiej (tzw. Y-DNA), wedle haplogrupy żeńskiej (mtDNA) oraz według autosomów (tzw. auDNA). Dlatego po konsultacji mailowej z dr Łukaszem Lubicz-Łapińskim, zajmującym się zawodowo tą tematyką, postanowiłem zlecić wykonanie takiego testu. Skorzystałem z oferty firmy „Genealogia Genetyczna” pana Lubicz-Łapińskiego, która zamawia testy w laboratorium National Geographic w Teksasie. Projekt badań DNA ludności prowadzony przez National Geographic nosi nazwę GENO 2.0. [1]
Wykupiłem pełen pakiet badań zwany „Family Finder”. W jego ramach uzyskuje się wyniki Y-DNA, mtDNA oraz wyniki autosomalne według regionów geograficznych świata oraz według podobieństwa do narodów. Koszt tego pakietu wyniósł mnie około 850 zł. Po dokonaniu opłaty przelewem, niedługo potem otrzymałem paczkę od dra Lubicz-Łapińskiego. Były w niej m.in. fiolki i patyczki do pobrania wymazów z policzka, a także katalog nt. wspomnianego projektu genetycznego. Fiolkę z dwoma wymazami wysłałem do USA dnia 26 stycznia 2019 r. Moje wyniki pojawiły się na stronie National Geographic dopiero 28 sierpnia 2019 r. W projekcie GENO 2.0 udział wzięło do tej pory ponad milion osób ze 140 państw świata.
Po tym krótkim wstępie przechodzę do omówienia wyników mojego testu DNA. Są one dość rozbudowane i składają się z kilku zakładek. Zacznę nie według kolejności zastosowanej przez National Geographic, ale od sprawy dla mnie najważniejszej – mianowicie od haplogrup i subkladów. Zakładka trzecia nosi nazwę „Your Deep Ancestry” i informuje jakie dana osoba posiada linie matczyne i ojcowskie. Jeśli chodzi o linię matczyną to wyszła mi gałąź T2b2. Jeśli zaś chodzi o linię ojcowską to jest nią gałąź CTS3402 należąca do słynnej haplogrupy R1a. Bardziej szczegółowych danych mi nie podano.
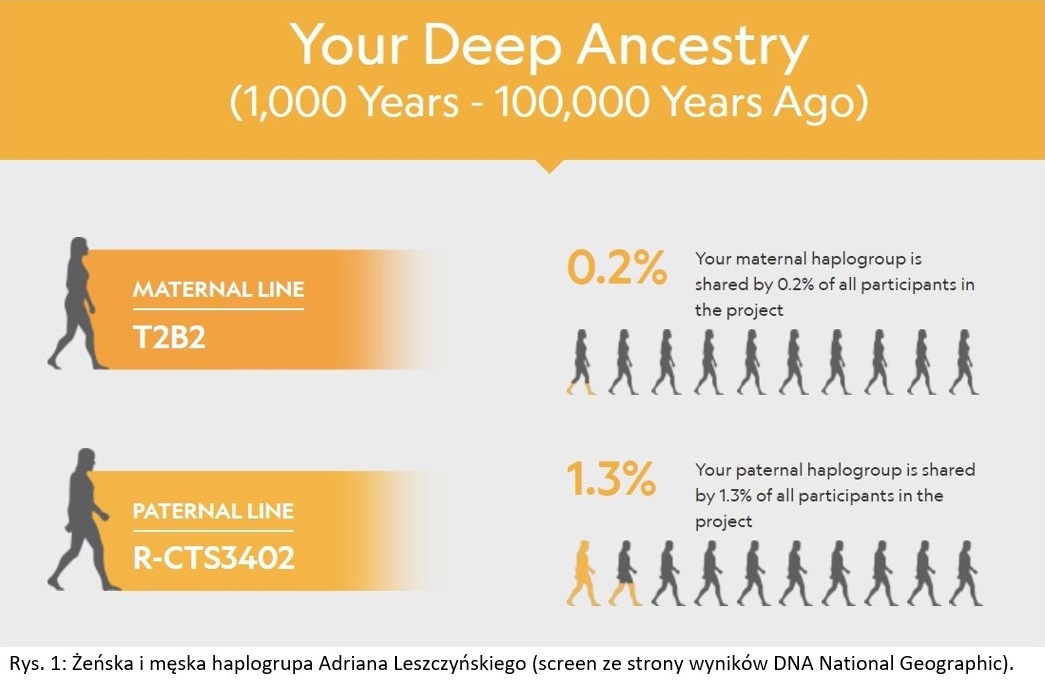
Powyższa grafika informuje, iż żeńską haplogrupę T2b2 posiada zaledwie 0,2% uczestników projektu GENO 2.0. Natomiast męską haplogrupę R1a-CTS3402 posiada również niewiele, bo jedynie 1,3% uczestników tego projektu.
Linia żeńska T2b2
T > T2 > T2b > T2b2
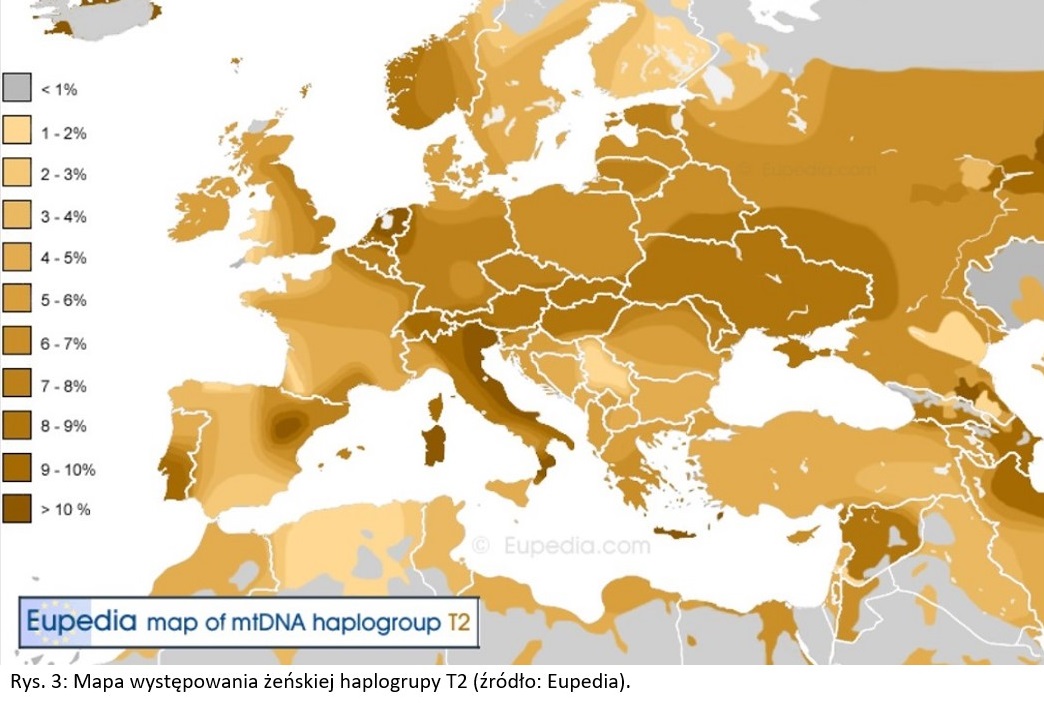
Linię żeńską, a więc mitochondrialne DNA (mtDNA) dziedziczy się po matce. Haplogrupa mojej matki to T2b2 będąca pochodną rodu T2, a wcześniej rodu T. Haplogrupa T2 osiąga najwyższe wartości wśród Udmurtów (24%), ugrofińskiego ludu z terenu Rosji. Wysoki odsetek posiadają też mieszkańcy Czeczenii, Inguszetii i Dagestanu (12,5%), rosyjskich republik na Kaukazie. Również inne narody i grupy etniczne z regionu Kaukazu charakteryzują się relatywnie wysokim odsetkiem tejże haplogrupy: Kumykowie (10%), Azerowie (9,5%) i Gruzini (9%). Wśród państw i regionów europejskich haplogrupa T2 najczęściej występuje w Holandii (12%), na Sardynii (10%), w Islandii (10%), Szwajcarii (9,5%), na Węgrzech (8,5%) oraz na Ukrainie (8,5%). [2]
Zgodnie z informacjami podanymi na stronie Eupedii, żeńska haplogrupa T odegrała ważną rolę w rozpowszechnianiu rolnictwa w całej Europie. Próbki T należące do T1a1’3, T2a1b1, T2b (w tym T2b3a i T2b23a), T2c (w tym T2c1d1), T2e i T2f znaleziono na stanowiskach kultury ceramiki wstęgowej rytej w Europie Środkowej i kultury Cucuteni-Trypole na Ukrainie. Zidentyfikowano też jedną próbkę T2 na stanowisku kultury ceramiki impresso-cardium w północno-wschodniej Hiszpanii. Naukowcy przetestowali próbki mtDNA z kultury grobów jamowych (ang. Yamna culture), domniemanej ojczyzny ludów indoeuropejskich i znaleźli gałęzie T2a1b w regionie środkowej Wołgi oraz w Bułgarii. Natomiast gałąź T1a znaleziono zarówno w środkowej części Ukrainy jak i w środkowym dorzeczu Wołgi. Częstotliwość występowania T1a i T2 w próbkach kultury grobów jamowych wynosiła 14,5% dla każdej z nich obu, co oznacza że procentowo były one wyższe niż w jakimkolwiek współczesnym państwie. W tak wysokich odsetkach haplogrupy te występują jedynie wśród narodu Udmurtów w regionie nadwołżańsko-uralskim w Rosji.
Haplogrupy T1 i T2 wykryto także wśród próbek z epoki brązu pobranych na stanowiskach kultury ceramiki sznurowej (T1a, T1a1’3, T2, T2b2b, T2b4f, T2c) i na stanowiskach kultury unietyckiej (T2b, T2c) w Europie Środkowej. Jak powszechnie już wiadomo, kultura ceramiki sznurowej związana była z ekspansją ludności o męskiej haplogrupie R1a ze stepów wschodnioeuropejskich, a kulturę unietycką wiąże się z przybyciem ludności o męskiej haplogrupie R1b na terytorium obecnych Niemiec. Współczesne linie matczyne T1 i T2 z Niemiec i Szwajcarii wykazują silną ciągłość z próbkami neolitycznymi z tych samych terytoriów. Kobiety o tych żeńskich haplogrupach zostały wchłonięte przez indoeuropejskich najeźdźców płci męskiej o haplogrupach R1a i R1b.
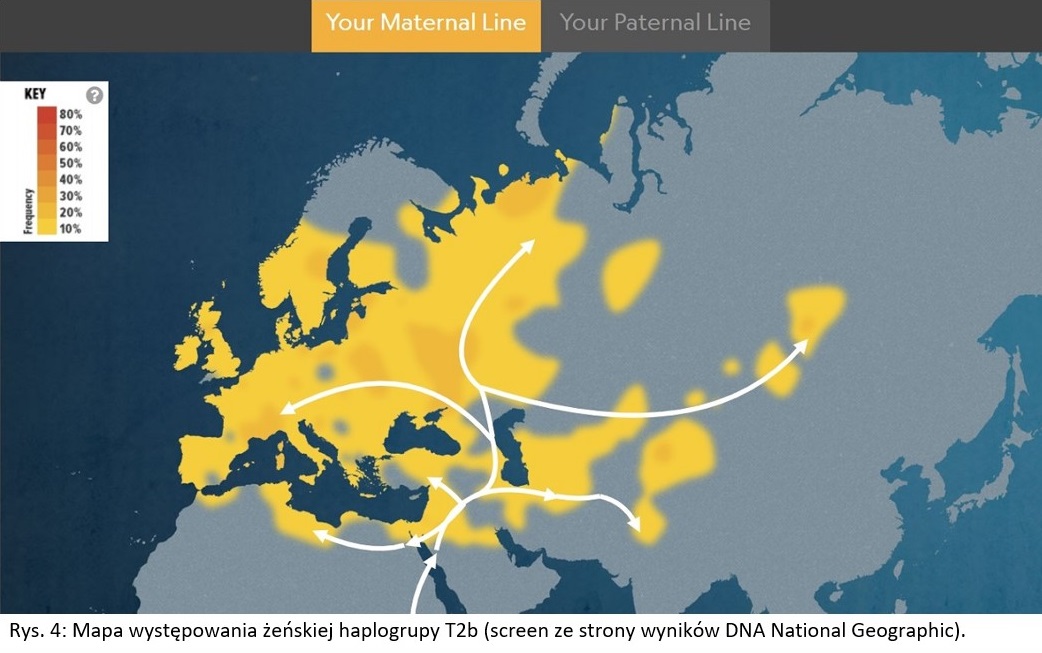
Według bazy National Geographic współcześnie gałąź T2b najczęściej występuje w Chorwacji (12%), Tunezji (9%), Grecji (5%), Iranie (4%), Armenii (3%). Wyniki te wedug Eupedii są nieco odmienne.
Poza Europą występowanie kladów pochodnych od T2 jest znacznie rzadsze. Jednakże zidentyfikowano je w środkowej i południowej Azji. Subklad T2b2 występuje w Turkmenistanie, Iranie i Indiach. Subklad T2b4 występuje w Uzbekistanie, subklad T2b11 znaleziono w regionie północnego Kaukazu, a T2b16 znaleziono w regionie nadwołżańsko-uralskim w Rosji oraz w Kazachstanie. Azjatyckie subklady T2b2 i T2b4 są tymi samymi, co te wyodrębione na stanowiskach kultury ceramiki sznurowej w Europie. Istnieje zatem wysokie prawdopodobieństwo, że występowanie żeńskich kladów T2b2 oraz T2b4 w Azji związane jest z migracją Pra-Indoeuropejczyków z Europy do Azji w okresie epoki brązu. Chodzi tu o migrację ludności, w której dominowała męska haplogrupa R1a. [3][4]

Linia męska R1a-CTS3402
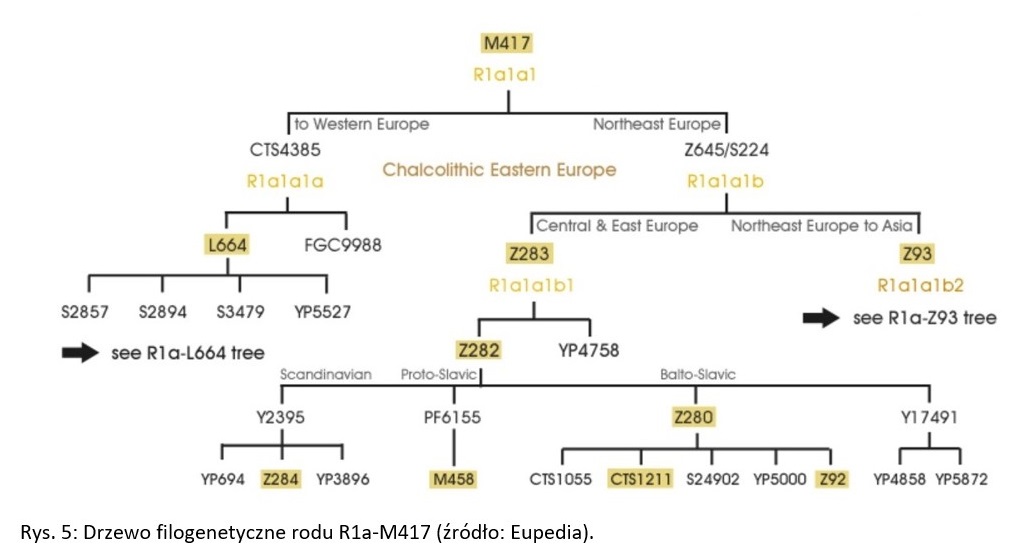
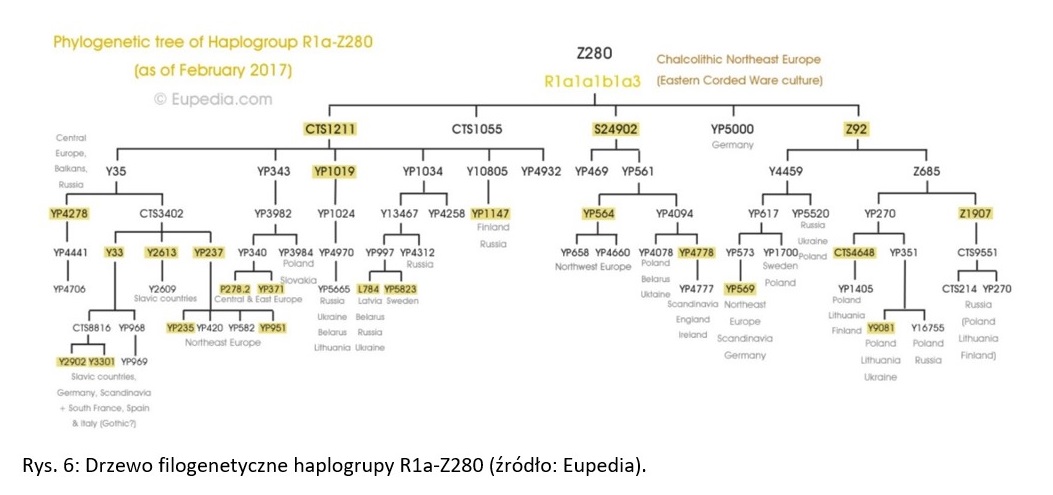
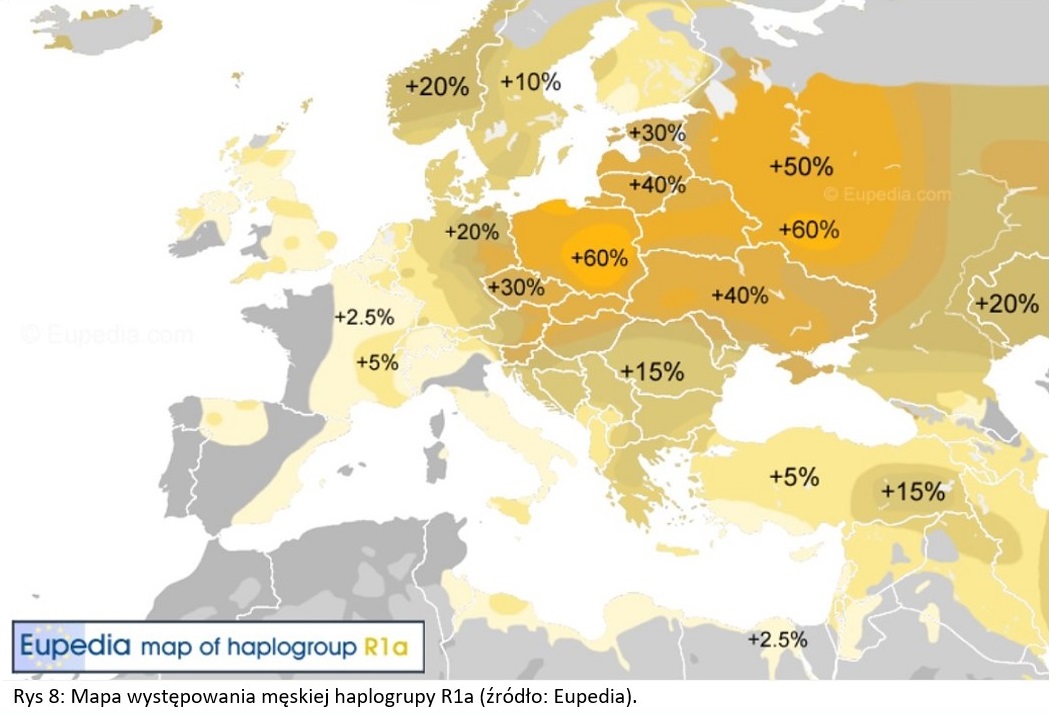
Męska haplogrupa dziedziczona jest dzięki chromosomowi Y. Ponieważ chromosom Y posiadają tylko mężczyźni, więc tylko oni dziedziczą po swych ojcach męską haplogrupę. R1a jest szeroko znaną haplogrupą wśród osób, które interesują się genetyką populacyjną, a nawet wśród osób, które z wiedzą o genetyce dopiero raczkują. Sam napisałem na jej temat wiele i odsyłam do moich starszych artykułów. [3][4] Jak wykazały badania DNA, ród genetyczny, z którego się wywodzę to także R1a. Haplogrupa ta dominuje w Europie Wschodniej, podczas gdy jej siostrzana haplogrupa R1b dominuje w Europie Zachodniej. Obie związane są historycznie z męską ludnością indoeuropejską, z tymże R1a związana jest bardziej z ludnością indoeuropejską satemową, a R1b bardziej z ludnością indoeuropejską kentumową. Z haplogrupy R1a wyrosła gałąź – najpierw M17, a następnie M417. Obecnie aż 99% ludności R1a należy do rodu M417. Niżej na drzewie filogenetycznym plasuje się klad Z645. Z niego wykształciły się dwa wielkie satemowe indoeuropejskie rody: Z93 oraz Z283. Ten pierwszy zrodził się najprawdopodobniej w Europie, a konkretnie na południoworosyjskich stepach i dał żywot ludności aryjskiej, która zaludniła Azję Środkową oraz Indie. Ten drugi dał początek ludności bałto-słowiańskiej, która zasiedliła Europę Środkową i Wschodnią, migrując również do Skandynawii i na Bałkany. Z kladu Z283 powstał Z282, a z niego wielki ród bałtosłowiański Z280, dominujący współcześnie wśród Słowian i silnie obecny u ludów bałtyckich. Od niego pochodzi typowo słowiańska gałąź CTS1211, a od niej Y35. Poniżej tej ostatniej jest CTS3402, do której przynależę. Bardziej szczegółowych wyników mi nie podano. [5]
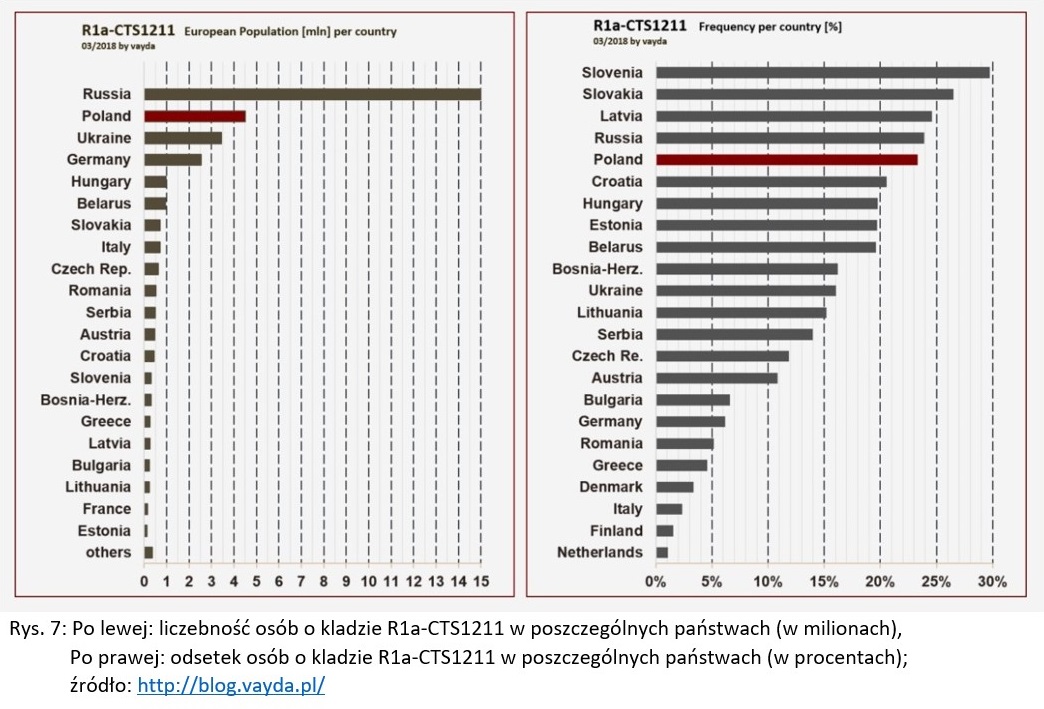
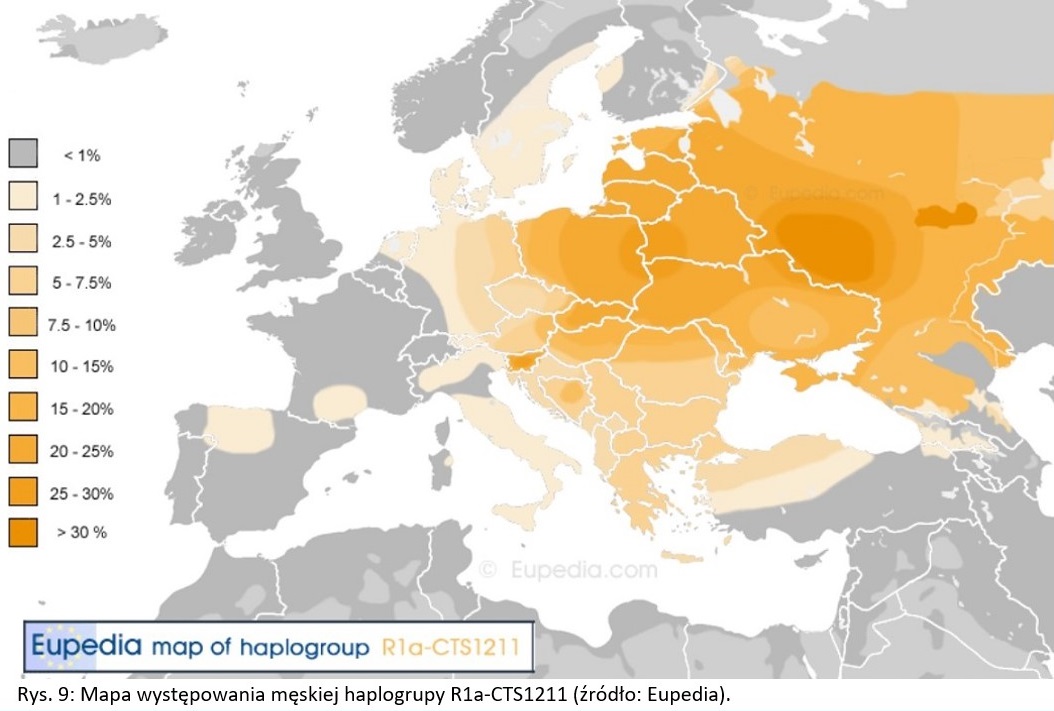
Wspomniana CTS1211 jest największą gałęzią rodu Z280. W swoich DNA subklad ten nosi ponad 34 miliony osób i występuje on powszechnie w krajach słowiańskich. Prawie połowa populacji CTS1211 mieszka w Rosji. Rozkład geograficzny tej mutacji pokazuje, że ludność prasłowiańska przemieszczała się od Renu po Ural na przestrzeni tysięcy lat. Częstotliwość występowania CTS1211 jest najwyższa w Słowenii (30,5%), na Słowacji (27,2%), na Łotwie (25,2%), w Rosji (24,3%), w Polsce (23,9%) i w Chorwacji (21,1%). Zatem blisko ¼ męskiej populacji Polaków przynależy do subkladu CTS1211. Jest to około 4,6 miliona osób. [6]
R > R1 > R1a > R1a-M417 > R1a-Z645 > R1a-Z283 > R1a-Z282 > R1a-Z280 > R1a-CTS1211 > R1a-Y35 > R1a-CTS3402
Wyniki autosomalne – podobieństwo do regionów świata
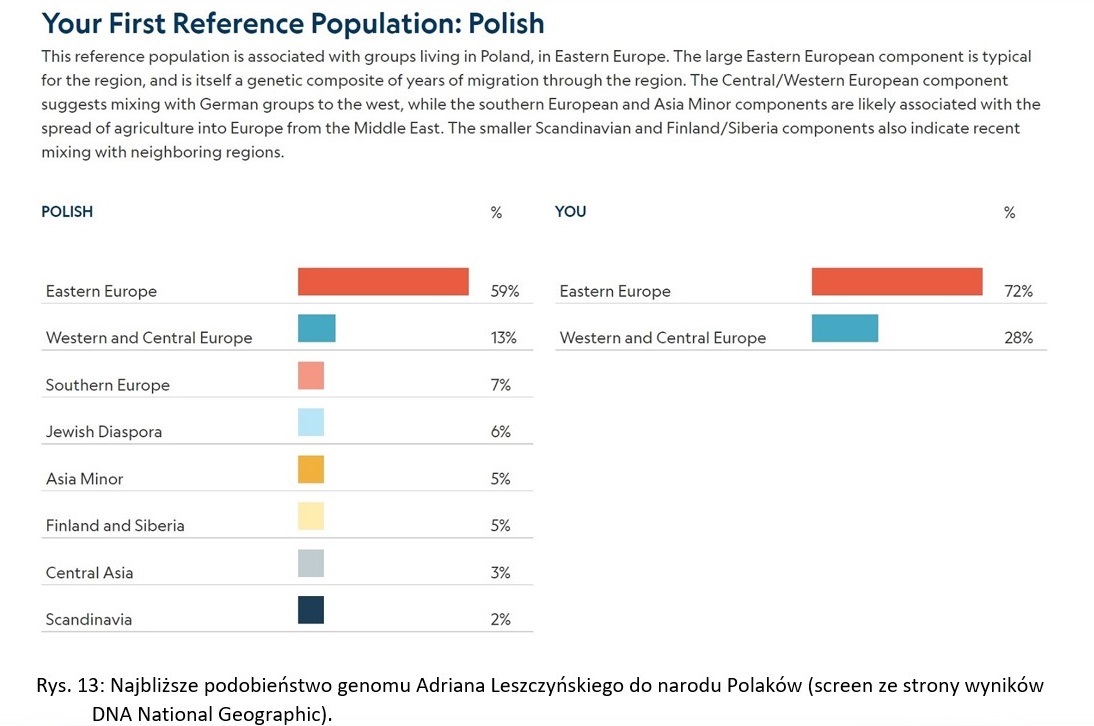
Genetyczne wyniki autosomalne pokazują podobieństwa genomu do współczesnych lub dawnych grup ludności. Mogą to być regionalne grupy ludności lub częściej narody. Pod względem statystycznym widać podobieństwo genomu danego człowieka do genomu jakiejś zbiorowości. Można też porównywać genom jednej zbiorowości do genomu innej zbiorowości. W badaniach tych pod uwagę bierze się nie tylko linię Y-DNA i linię mtDNA, ale wszelkie linie boczne. W projekcie GENO 2.0 najczęściej bierze się pod uwagę linie sześciu pokoleń wstecz. Każdy człowiek ma dwóch rodziców. Rodzice to jedno pokolenie wstecz. Ale dziadków, którzy są dwa pokolenia wstecz jest już czterech, a więc 2 razy więcej niż rodziców. Pradziadków jest już ośmiu (3 pokolenia wstecz). Przodków 6 pokoleń wstecz jest już 64. Tym samym badania autosomalne dotyczą w tym przypadku co najmniej 64 linii pochodzenia danego osobnika. Na podstawie statystycznych porównań tych co najmniej 64 linii przodków z genomami współczesnych narodów zgromadzonych w bazie National Geographic wychodzi, że 72% mojego genomu najbliższe jest genomowi osób z Europy Wschodniej, a 28% najbliższe jest genomowi osób z Europy Centralnej i Zachodniej. Polska według tego projektu, zgodnie z amerykańskim punktem widzenia, należy właśnie do Europy Wschodniej. W badaniu tym świat podzielono na 18 regionów, z których mój genom dzieli podobieństwo tylko z dwoma regionami – oboma z Europy. Przyznać muszę, że ten wynik trochę mnie zdziwił, gdyż przewidywałem w moich wynikach domieszki azjatyckiej krwi. Z tych badań ta spodziewana domieszka mi nie wyszła. Wyszło za to, że jestem stuprocentowym Europejczykiem.


Wyniki autosomalne – podobieństwo do narodów
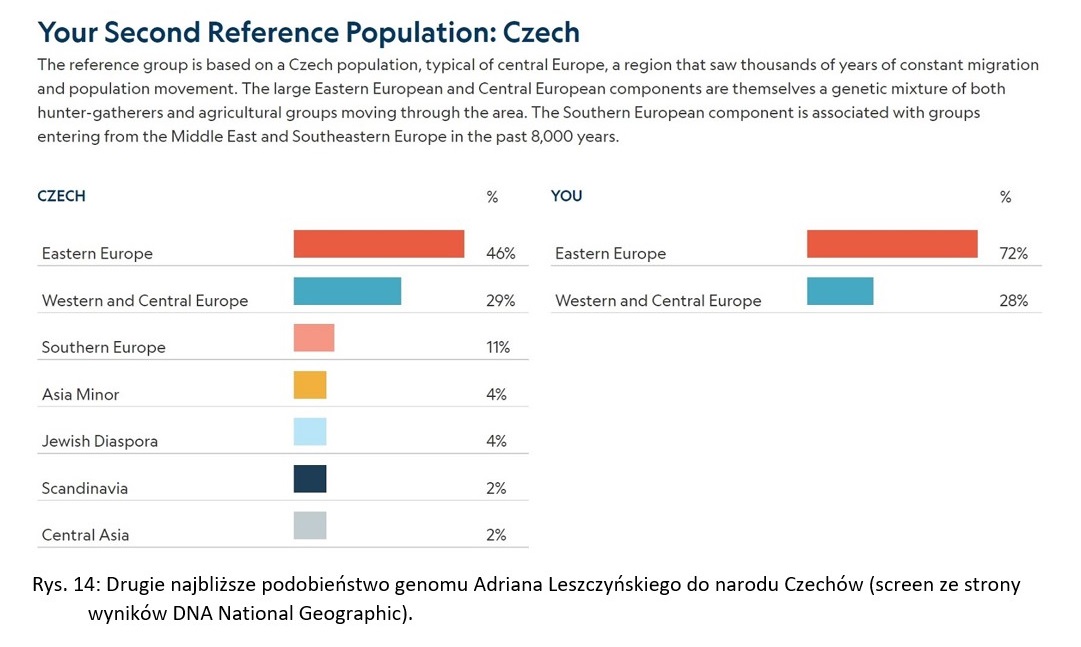
Jeśli zaś chodzi o podobieństwo mojego genomu do poszczególnych narodów, to najbliżej mi jest do Polaków. Na drugim miejscu pod względem mojego genetycznego podobieństwa plasują się Czesi. W tym miejscu zaskoczę trochę moich Czytelników twierdząc, że tu także spodziewałem się nieco innych wyników. Ze względu na łemkowskie pochodzenie mojej matki oraz ze względu na kresowe korzenie mojego ojca, wydawało mi się, że pod tym względem na drugim miejscu znajdą się Ukraińcy. Mało tego! Nie zdziwiłbym się, gdyby wyszło, że mój genom bliższy jest Ukraińcom niż Polakom. Ukraińcy zaś, nie znaleźli się nawet na drugim miejscu w tym zestawieniu. Natomiast bardzo zdziwiło mnie moje podobieństwo do Czechów. Prędzej spodziewałbym się podobieństwa do Słowaków. Na Słowacji bowiem żyje sporo osób łemkowskiego i rusińskiego pochodzenia, więc moje podobieństwo do mieszkańców Słowacji byłoby całkiem realne. Moja matka urodziła się w łemkowskiej wsi położonej zaledwie 5 km od słowackiej granicy, a tamtejsi polscy Łemkowie dość często wchodzili w związki małżeńskie z Łemkami mieszkającymi po stronie słowackiej. Z wiadomych mi informacji, z Czechami zaś nie mam żadnych koligacji. Przynajmniej nic mi o tym nie wiadomo. Moje pochodzenie zarówno od strony matki jak i od strony ojca miałem okazję opisać w dwóch moich fotoreportażach. [7][8]
Wspólne pochodzenie z geniuszami
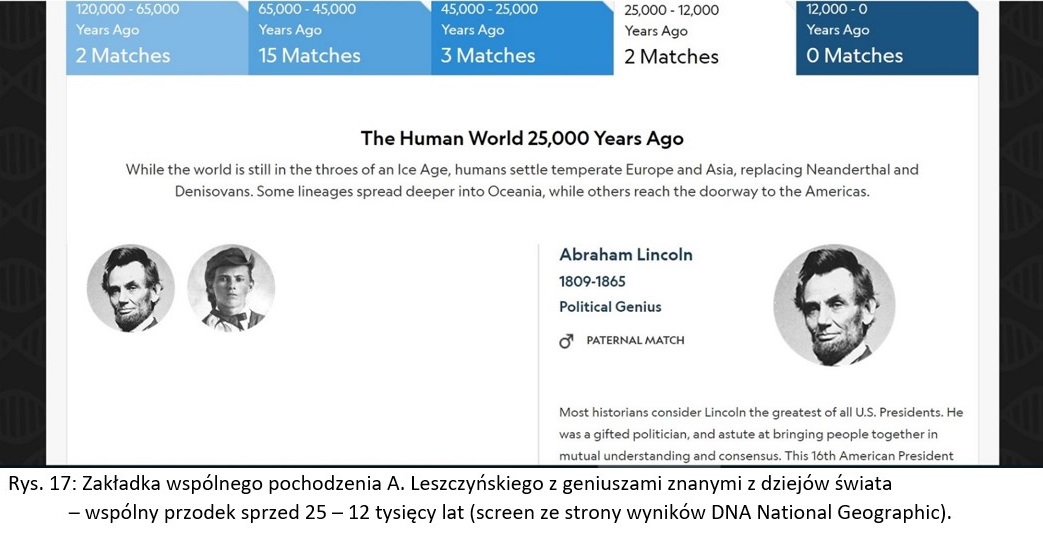
Pierwsza zakładka projektu GENO 2.0 nosi nazwę „Your Genius Matches” i pokazuje moje wspólne pochodzenia z osobami słynnymi z dziejów świata, z tzw. geniuszami. Okazało się, że najbliższych przodków dzielę z takimi osobami jak Abraham Lincoln (prezydent Stanów Zjednoczonych z czasów wojny secesyjnej) i Jesse James (XIX-wieczny amerykański przestępca, rewolwerowiec). Wspólnych przodków miałem z nimi około 12.000 – 25.000 lat temu. Z amerykańskim prezydentem miałem wspólnego przodka w linii ojcowskiej, natomiast ze słynnym rozrabiaką i rewolwerowcem w linii matczynej.

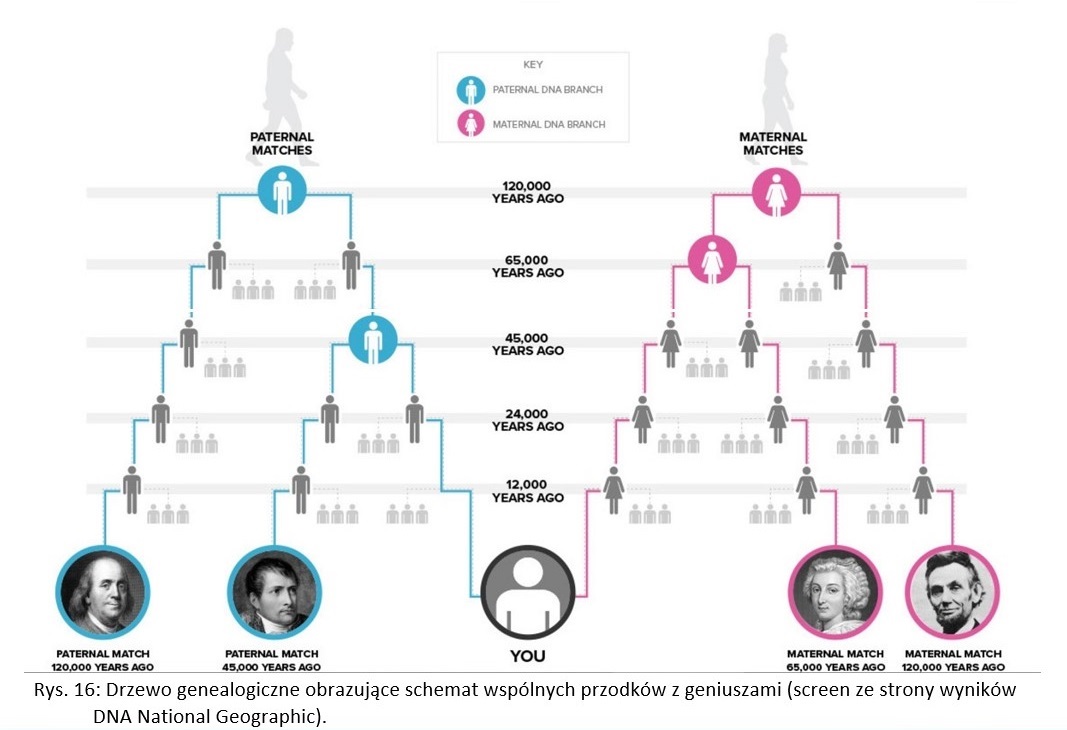
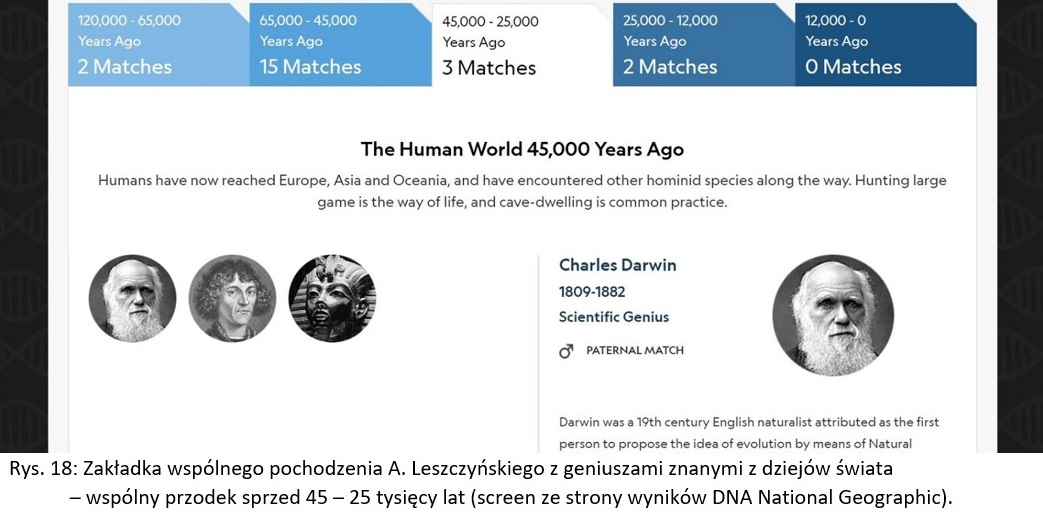
Z trzema innymi słynnymi geniuszami miałem wspólnego przodka 45.000 – 25.000 lat temu. Tymi osobami są: Karol Darwin, Mikołaj Kopernik oraz faraon Tutanchamon. Z całą tą trójką geniuszy miałem wspólnych przodków w linii ojcowskiej.
65 – 45 tysięcy lat temu żyli moi przodkowie, którzy także byli przodkami słynnych osób. W tym zestawieniu ponownie pojawia się amerykański prezydent Abraham Lincoln oraz Mikołaj Kopernik. Tym razem jednak mam z nimi wspólnego przodka, ale w linii od strony mojej matki. Przodków sprzed 65 – 45 tysięcy lat w linii matczynej dzielę ponadto z takimi osobistościami jak: Francesco Petrarca, angielska królowa Wiktoria, Beniamin Franklin, królowa Francji Maria Antonina, Napoleon Bonaparte oraz królowa Czech i Węgier Maria Teresa Habsburg.
Natomiast w linii ojcowskiej wspólnego przodka 65 – 45 tysięcy lat temu miałem z następującymi postaciami: Czyngis-chan, Lew Tołstoj, Tomasz Jefferson, Nikola Tesla, Alexander Hamilton (amerykański polityk) i Marcin Luter.
Z angielskim królem Ryszardem III dzielę wspólnych przodków w wyżej wspomnianym okresie zarówno od strony mojej matki jak i od strony mojego ojca.

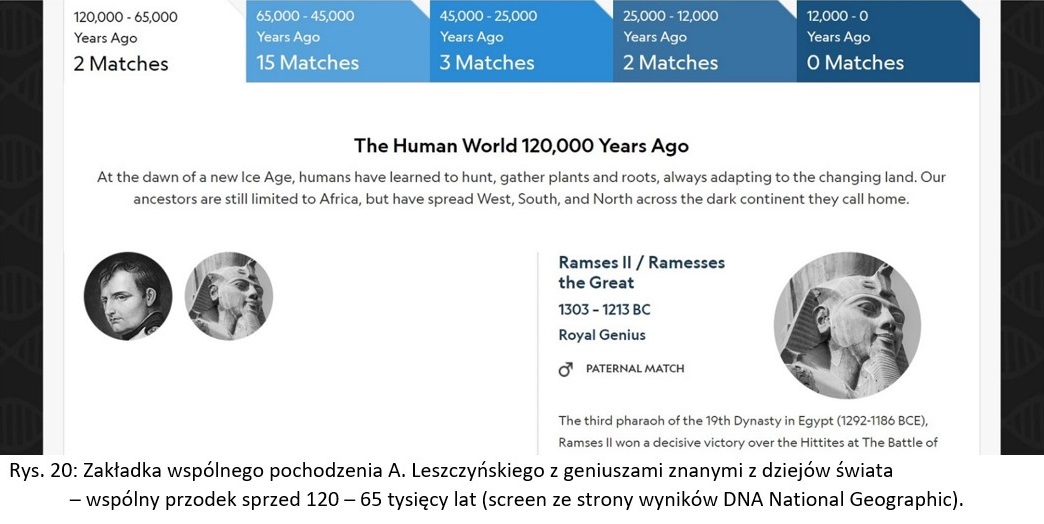
W zestawieniu wspólnych przodków sprzed 120 – 65 tysięcy lat ponownie pojawia się Napoleon Bonaparte. Tym razem mam z nim wspólnego przodka w jednej z linii ojcowskich. Wspólnego przodka z tego okresu dzielę także z faraonem Ramzesem II i także jest to jedna z linii od strony mojego ojca.
Pochodzenie od różnych podgatunków człowieka
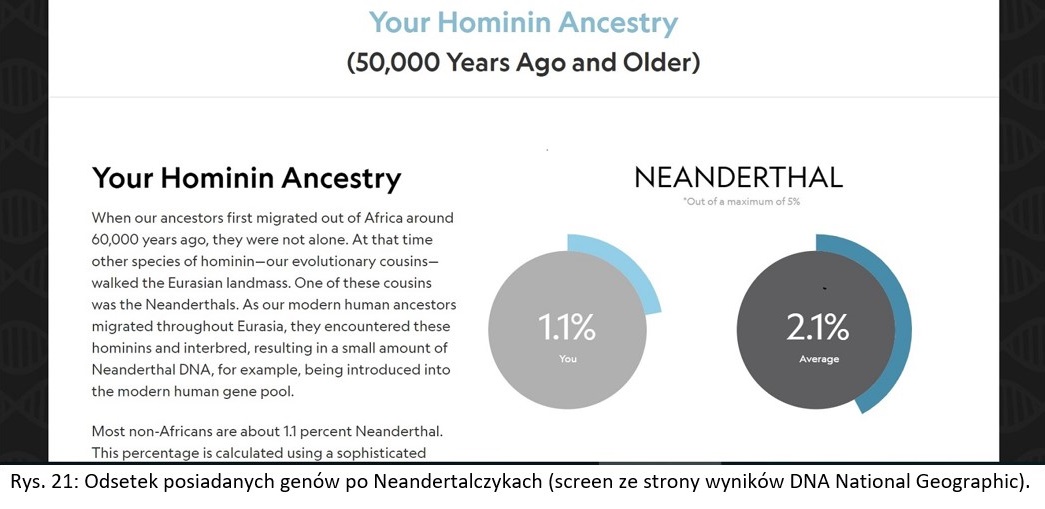
Ostatnia zakładka projektu GENO 2.0 pokazuje pochodzenie badanej osoby od różnych podgatunków człowieka. Według informacji National Geographic współczesna ludność świata posiada około 2,1% pochodzenia od wymarłego podgatunku człowieka, zwanego Neandertalczykiem (łac. Homo sapiens neanderthalensis). Oznacza to, że każdy współcześnie żyjący człowiek na Ziemi posiada około 2,1% genów pochodzących od Neandertalczyka. W przypadku ludności poza-afrykańskiej odsetek ten spada do około 1,1%. Taki też wynik wyszedł dla mojego DNA.
PRZYPISY:
[1] – GENO 2.0 National Geographic; https://genographic.nationalgeographic.com/
[2] – Haplogroup T (mtDNA); Eupedia; https://www.eupedia.com/europe/Haplogroup_T_mtDNA.shtml
[3] – Adrian Leszczyński; Krótka historia rodu genetycznego R1a; Białczyński 17.01.2017; https://bialczynski.pl/2017/01/17/adrian-leszczynski-krotka-historia-rodu-genetycznego-r1a-2/
– Skribh 17.01.2017; https://skribh.wordpress.com/tag/krotka-historia-rodu-genetycznego-r1a/
[4] – Adrian Leszczyński; Języki słowiańskie a haplogrupa R1a1; Taraka 16.03.2016:
http://www.taraka.pl/jezyki_slowianskie_a_haplogrupa_R1a1
[5] – Haplogroup R1a (Y-DNA); Eupedia; https://www.eupedia.com/europe/Haplogroup_R1a_Y-DNA.shtml
[6] – Vayda; R1a-Z280 [#17; 03/2018]; http://blog.vayda.pl/r1a/r1a-cts1211/
[7] – Adrian Leszczyński; Fotoreportaż z krainy Słowian – Łemkowszczyzna; Białczyński 21.04.2018; https://bialczynski.pl/2018/04/21/adrian-leszczynski-fotoreportaz-lemkowszczyzna/
[8] – Adrian Leszczyński; Fotoreportaż z Kresów – dawne województwo stanisławowskie; 12.05.2019; https://bialczynski.pl/2019/05/12/adrian-leszczynski-fotoreportaz-z-kresow-dawne-wojewodztwo-stanislawowskie/
KONIEC
Adrian Leszczyński
aleszczynski@interia.pl
